
脳神経系WG
大規模数理モデル構築を目的とした共有モデル開発
プラットフォーム:PLATO
① 理化学研究所 次世代計算科学研究開発プログラム
② 理化学研究所 脳科学総合研究センター
(左から)稲垣 圭一郎①、観音 隆幸②、Nilton L. Kamiji②、槇村 浩司②、臼井 支朗①②

脳により営まれている情報処理の解明は、自然科学における最難題に位置付けられています。
脳では、約1000億個の神経細胞が約10兆にのぼるシナプスと呼ばれる結合により、ネットワークを形成しています。
視覚や聴覚など外界から得られる様々な情報は、こうした脳のネットワークで柔軟かつ適切に処理されていると考えられています。
こうした脳における複雑な情報処理は、主として電気生理実験により、その機能が明らかにされつつあります。
また、近年の計算科学の飛躍的な進歩に伴い、脳の一部を詳細に数理モデルとして構築しシミュレーションを行うことで、計算科学の立場から脳機能を解明しようとする研究も行われています。
しかしながら、未だに脳全体を一つの大規模数理モデルとして記述しシミュレーションすることは簡単ではありません。
脳の大規模モデルの構築には、膨大な知識や技術が必要であることから、一人の研究者単独で成し遂げることは困難であり、むしろ多くの研究者の共同作業により、得られた知識を集約・蓄積し、その本質をコンピュータ上で動作する大規模モデルとして統合していくことが重要であると考えられます。
我々は、脳の大規模モデルを構築する新しいアプローチとして、従来の生理学的ならびに計算論的研究によって得られた脳の各部位に関する知識や数理モデルを統合して大規模化する方法を研究しています。
我々のアプローチでは、データベースサーバ(エール大学のModelDBやNeuroinformatics Japan Nodeの各プラットフォームなど)に登録されている数理モデルや開発に必要なデータの収集・管理ツール、シミュレータなどのモデル開発環境とシミュレーションサーバ、および結果の描画ツールからなる統合モデル開発環境の構築を進めています(図1)。
特に、複数のモデルを簡単に結合できるように、モデルの入出力を標準化してデータの受け渡しを可能にするデータフォーマットとその利用支援ライブラリについても研究・開発しています。
標準化されたフォーマットは、プログラミング言語を問わない仕組みになっており、例えば、C言語で作られた数理モデルとPythonで作られた数理モデルというように異なる言語で構築されているモデルの結合も可能にしています。
さらに、モデル入出力が標準化されていることから、大規模モデルに統合したモデルの変更・更新、ならびに新しい数理モデルの統合に対してもプラグイン形式で対応が可能です。
標準化されたフォーマットにより結合されたモデルのシミュレーションでは、Agentというシステムによって、統合した各数理モデルを一つの大規模モデルとして同時並列に実行することを可能にしています。
実行時には各モデルの実行ステップ間隔を考慮した実行管理により、モデル間のデータ通信についても自動的に調整されます。
こうした標準フォーマットの利用やAgentによるモデル実行に関するライブラリを提供することで、モデルの作成者は独自のコードをほとんど変更することなく他のモデルと結合可能な数理モデルの開発が可能になるものと考えています。
我々は、脳機能の一つである視覚、つまり「ものを見る」ことに焦点をあて、視覚に関する脳の大規模モデル構築を前述のアプローチにより進めています。
ヒトの視覚は、眼球、眼光学系、網膜、脳皮質により構成されており、外界情報が、これらの部位で多階層的に処理されることで、物の認識や視線の移動といった機能が実現されています。
こうした脳の各部位に関する数理モデルを構築し、前述の方法で統合しシミュレーションを行うことで、ヒトがものを見ている際の脳内情報処理の可視化を目指しています(図2)。
今後、次世代スーパーコンピューターを用いて、“視覚”に関わる様々な研究者との共同作業によって、視覚に関わる全脳部位を詳細に記述し、さらに、我々の提案するアプローチによってそれらを統合し、一つの大規模視覚系モデルとして計算機シミュレーションを行うことで、脳内で行われている視覚情報処理の解明が促進されていくものと考えています。
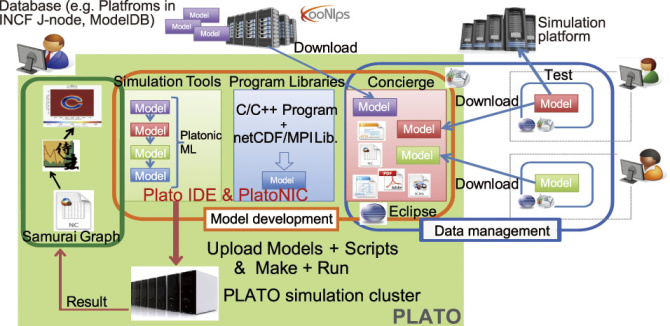
図1: PLATOシステム環境 PLATOはデータ管理ツール(http://concierge.sourceforge.jp/)、モデル開発環境、シミュレーションサーバ、結果の描画ツールの4つの環境から構成されている。
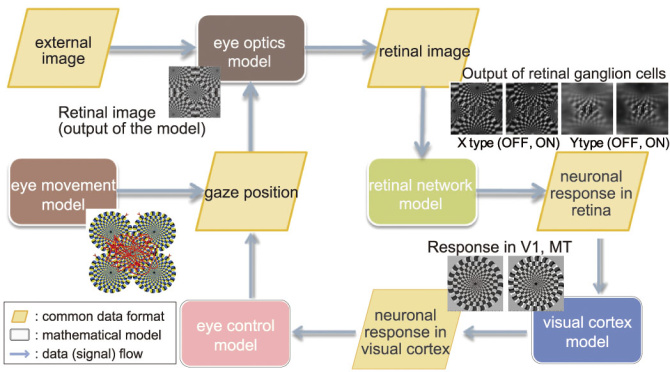
図2:大規模視覚モデルの構成図眼球運動、眼光学、網膜、皮質を構成した視覚系の数理モデルとシミュレーション時の各モデル出力のサンプル画像。各脳部位の数理モデルは、それぞれ個別のモデルとして記述されており、標準化されたフォーマット(Common dataformat)により各モデルが結合されています。
BioSupercomputing Newsletter Vol.4
- SPECIAL INTERVIEW
- 経験重視の観察型医療から予測型医療への転換を図り、理論医学の基盤を構築するために
東海大学医学部内科学系(循環器内科)教授
東海大学医学研究科バイオ研究医療センター代謝疾患研究センター長
東海大学総合医学研究所代謝システム医学部門長 後藤 信哉 - シミュレーション科学の活用で栄養学や健康管理の新たな可能性が拓かれることに期待
味の素株式会社 ヘルスインフォマティクス班
グループ・エクゼクティブ・プロフェッショナル員 安東 敏彦
- 研究報告
- 多剤排出トランスポーターの機能を粗視化分子シミュレーションで実証(分子スケールWG)
京都大学理学研究科 高田 彰二/姚 新秋/検崎 博生 - 時空間を考慮した細胞シミュレーション(細胞スケールWG)
理化学研究所 次世代計算科学研究開発プログラム 須永泰弘 - 強力集束超音波による低侵襲治療のためのHIFUシミュレータの開発(臓器全身スケールWG)
理化学研究所 VCADシステム研究プログラム 沖田 浩平 - 大規模数理モデル構築を目的とした共有モデル開発プラットフォーム:PLATO(脳神経系WG)
①理化学研究所 次世代計算科学研究開発プログラム
②理化学研究所 脳科学総合研究センター
稲垣 圭一郎 ①/観音 隆幸 ②/ Nilton L. Kamiji ②/槇村 浩司 ②/臼井 支朗 ①②
- 報告
- BMB2010(第33回日本分子生物学会年会 第83回日本生化学会大会、合同大会)におけるワークショップ開催報告
- 生命体統合シミュレーション ウィンタースクール2011
理化学研究所 次世代計算科学研究開発プログラム 石峯 康浩(臓器全身スケールWG)
東京大学医科学研究所 浦久保 秀俊(脳神経系WG)
理化学研究所 次世代計算科学研究開発プログラム 須永 泰弘(細胞スケールWG)
理化学研究所 次世代計算科学研究開発プログラム 舛本 現(開発・高度化T)
理化学研究所 次世代計算科学研究開発プログラム 三沢 計治(データ解析WG)
理化学研究所 次世代計算科学研究開発プログラム 宮下 尚之(分子スケールWG)
