
分子スケールWG
QM/MD/CGMのマルチスケール分子シミュレーションの実現
大阪大学蛋白質研究所
(左から)米澤康滋、山中秀介、下山紘充、山﨑秀樹、中村春木
理化学研究所次世代計算科学研究開発プログラム
福田育夫

私たちのグループは、蛋白質や核酸が生体内でその機能を果たす仕組みを、分子シミュレーションを使って、分子、原子、電子にわたるマルチレベルで解明しようとしています。現実の生命体分子は室温で熱的に揺らぐ動作をしていて、その揺らぎが分子の機能に大きな影響を与えています。私たちの目的は、その熱揺らぎをシミュレーションしつつ、電子状態まで含めて、生命体分子の働く仕組みをできるだけリアルに解明することにあります。
その実現のために、私たちは個別に開発されてきた量子化学計算(QM)/分子動力学計算(MD)を連成させた(QM/MD)マルチスケールシミュレーションプログラム Platypus (PLATform for dYnamic Protein Unified Simulation) を開発しています。電子状態を計算する量子化学は、酵素機能をシミュレーションで取り扱う時に必須の計算手法であり、ここではHartree-Fock (HF) 法、密度汎関数法 (DFT)、CASSCF, CISによる計算が可能です。分子動力学法は、分子が熱的に揺らぐ姿を再現するシミュレーション方法です。Platypusはオリジナルなコードによって超並列を実現するプログラムの総体ですが、これまでの開発によってHF, DFTの演算とも8192並列までの加速性能を発揮しており、特に1000 〜 2000並列程度までは良いスケーラビリティーを示しています。Platypusでは、これらに加えて独自に開発した粗視化モデル(CGM)によるMD計算も搭載し、QM/MD/CGMのマルチスケールの分子シミュレーションが実施できます。さらに私たちは分子動力学との連携を取りながらQM/MD計算による効率良いサンプリングを行うアルゴリズムも開発しています。
Platypusの応用例の一つとして、生体内で様々な生命活動のシグナル分子制御に関係しているプロリン残基シス-トランス異性化反応機構の研究を紹介します。水中でのプロリンを含む小さなペプチドの研究(文献1)では、シス状態とトランス状態の間にある遷移状態が従来知られているピラミッド型以外に逆ピラミッド型を成し、かつこの二つの型の間で揺らいでいることを初めて示しました(図1)。またプロリン残基のシス-トランス異性化を促進するPin1異性化酵素の研究からは、酵素活性部位にプロリンを含むペプチドが結合することで生じるひずみが遷移状態のエネルギーを安定化して異性化を促進する機構を捉えることに成功しています。
分子動力学シミュレーションでは、主に分子間で化学結合によらない原子間の長距離力の計算時間が大部分を占めています。この部分を並列化することで計算時間を大幅に短縮することが可能となります。Ewald法が最も良く用いられている一般的な方法ですが、このEwald法は非常に大きな系の並列化には向いていないことが知られています。そこで私たちは、最近Wolfらによって開発された長距離力ポテンシャルを改良し、矛盾無くかつ安定なシミュレーションができるようにしたポテンシャルを用いるForce Switching-Wolf法(FSw-Wolf法)を開発し、Ewald法の代わりとして利用する研究を進めています。FSw-Wolf法はEwald法に匹敵する精度の力の計算を実現できることを、私たちは、融解ナトリウム塩(文献2、図2)と水中の短いペプチドのシミュレーションで示すことに成功しています。FSw-Wolf法は非常に簡便なアルゴリズムであるとともに比較的短距離の原子間の力のみを考慮するだけなので、大規模な並列化分子動力学シミュレーションに大変良く適している方法で、他のアルゴリズムと併用することで高速な超並列計算を実現できるカギとなる方法であると考えています。
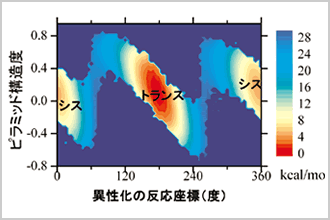
図1:QM/MDマルチスケールシミュレーションで解明された、プロリン残基を含むペプチドの異性化自由エネルギー地形。シスとトランス状態の間に揺らぎの大きな遷移状態が認められる。 |
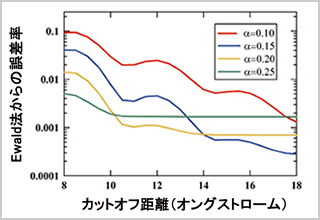
図2:FSw-Wolf法とEwald法のエネルギー誤差を、FSw-Wolf法のカットオフ距離とパラメータαの関数としてプロットした。 FSw-Wolf法がEwald法に大変近いエネルギーを再現できることを示している。 |
文献1. Yonezawa Y., Nakata K., Sakakura, K, Takada T., Nakamura H., J. Am. Chem. Soc. 131 (12), 4535–4540,
2009.
文献2. Fukuda I., Yonezawa Y., Nakamura H., J. Phys. Soc. Jpn. 77 (11), 114301, 2008.
BioSupercomputing Newsletter Vol.3
- SPECIAL INTERVIEW
- さまざまな最先端研究基盤を統合して活かすためにも
スーパーコンピュータの果たす役割は大きい
持田製薬株式会社 医薬開発本部 専任主事 東北大学 客員教授 西島 和三 - 超音波治療の推進および治療機器開発に欠かせない生体の音響的シミュレーション研究
東京大学大学院 工学系研究科 機械工学専攻 特任研究員 佐々木 明
- 研究報告
- QM/MD/CGMのマルチスケール分子シミュレーションの実現(分子スケールWG)
大阪大学蛋白質研究所 米澤康滋/山中秀介/下山紘充/山﨑秀樹/中村春木
理化学研究所 次世代計算科学研究開発プログラム 福田育夫 - 細胞レベルの精緻な代謝モデルを用いた
肝臓シミュレータの開発と実証に向けて(細胞スケールWG)
慶應義塾大学医学部 谷内江 綾子 - MEGADOCKによる網羅的タンパク質間相互作用予測(データ解析融合WG)
東京工業大学大学院情報理工学研究科 秋山 泰/松崎 由理/内古閑 伸之/大上 雅史 - 昆虫嗅覚系全脳シミュレーション(脳神経系WG)
東京大学先端科学技術研究センター 加沢 知毅
- 報告
- 生命体統合シミュレーションサマースクール2010を開催
理化学研究所次世代計算科学研究開発プログラム 石峯 康浩(臓器全身スケールWG)
東京大学医科学研究所 島村 徹平(データ解析WG)
理化学研究所次世代計算科学研究開発プログラム 須永 泰弘(細胞スケールWG)
京都大学大学院情報学研究科 本田 直樹(脳神経系WG)
理化学研究所次世代計算科学研究開発プログラム 舛本 現(開発・高度化T)
理化学研究所次世代計算科学研究開発プログラム 松永 康佑(分子スケールWG) - 生命体統合シミュレーション・サマースクール2010へ参加して
東京大学大学院理学系研究科博士課程1年 齊藤 健
