 HPCI戦略プログラム 分野1 予測する生命科学・医療および創薬基盤
HPCI戦略プログラム 分野1 予測する生命科学・医療および創薬基盤
課題4 大規模生命データ解析
最先端シークエンサーによる大規模データを
「京」で解析し生命プログラムとその多様性の理解を進める

東京大学医科学研究所ヒトゲノム解析センター 教授
宮野 悟
(課題4 代表)
●大規模データ解析で生命システムを明らかに
課題4「大規模生命データ解析」は、「京」を中核とした高性能計算施設(HPCI)に最適化した最先端・大規模シークエンスデータの解析基盤を整備した上で、がんや細胞分化のプロセスに現れる生命プログラムをシステムとして捉え、その複雑性や多様性をゲノムによって理解する研究を進めるとともに、ゲノムを基軸とした生体分子ネットワーク解析研究を実施しています。そして、こうした研究によって、薬効・副作用の予測、毒性の原因の推定、オーダーメード投薬、予後予測などへの応用に貢献することをめざしています。
課題の名称にある「生命データ」は、いうまでもなくゲノム、エピゲノム(DNA修飾の総体)、トランスクリプトーム(転写産物の総体)など、ゲノムを基軸とした大量データをさしています。これらは、生命プログラムを理解するための重要なデータです。
その重要性を理解してもらうための一例として、私たちはなぜがんになるのかを考えてみましょう。親から受けた遺伝的要因、環境的要因によるDNAの修飾(エピゲノム)、腫瘍細胞に蓄積した遺伝子変異──これらが複雑に組み合わさって引き起こされる生命システムの何らかの異常、それががんです。システム異常が起こると、本来異常が起きないように自滅する仕組み(アポトーシス)が機能しなかったり、増殖を止める外部からの命令が機能せず、自分自身で増殖命令を出したりといったことが起きます。さらにがん細胞は、血管内皮細胞や免疫炎症細胞などの正常細胞とシェークハンドしながら薬剤耐性を獲得したり、浸潤と転移でどこへでも広がっていくなど、まさに時空間で進化する複雑で異常な細胞集団なのです。こうしたがんの悪性度や治療応答性、副作用の出やすさなどを規定しているのがゲノムであり、システム異常を明らかにするためには、ゲノム、エピゲノム、トランスクリプトーム、さらにはプロテオーム(タンパク質の総体)、メタボローム(代謝産物の総体)、インタラクトーム(相互作用の総体)などを見ていかなければなりません。しかし、それだけで治療法や病気の状態が理解できるというわけではありません。がんの多様性・複雑性やダイナミズムを解き明かすためには、数学とスーパーコンピュータを駆使した大規模データ解析と数理モデリングによって、システムを理解することが重要なのです。
●「京」による数理解析で生命システム理解にブレークスルーを
2003年、13年の歳月と1千億円という費用をつぎ込んだ「ヒトゲノム計画」によって、ヒトのゲノムが解読されました。この年に、米国国立衛生研究所(NIH)は、ヒトゲノム解読後のロードマップを出しましたが、そのなかに「Biology is changing fast into a Science of Information Management.」という一文があります。当時、多くの日本の研究者たちは、驚きとともに「言い過ぎでは」と思ったものですが、10年過ぎた今、誰もがそうなりつつあることを実感しています。さらに米国を中心に、2003年から個人の違いをDNAレベルで解明する国際ハプマップ計画が行われ、ヒトの病気や薬剤応答に関わる遺伝子を効率よく見つけるための基盤ができました。2008年からは主要ながんのゲノム異常カタログを作成する国際がんゲノム計画が始まり、2万5千人のがんサンプルと正常細胞、合わせて5万人分の全ゲノム情報が解析されています。一方で、2008年に大統領に就任したバラク・オバマ氏は、上院議員時代に「ゲノムと個別化医療法案」を提出し、2009年には「遺伝子差別禁止法」が成立するなど、米国のゲノム情報を基盤とする医療ヘルスケア戦略は、着々と進められてきたわけです。また、NIHは早くも2004年から「1千ドルゲノム計画」(1千ドルでヒト一人のゲノムを読めるように解析のコストダウンをめざす計画)に予算を付け始め、シークエンス技術の発展とその実用化が進み、今まさにバイオロジーは、大量のゲノムデータをどう解析し、解釈していくかというサイエンスになろうとしています。
シークエンス技術については、今日その高度化が急速に進んでいます。これまでの光シークエンス技術を用いた次世代シークエンサーは、装置そのものが高価である上、非常に高価な蛍光試薬が必要であることやリード長も限られているといった課題がありました。しかし現在、コストの安いシリコンチップを使う半導体シークエンサーのような最先端シークエンサーの開発が進み、2013年には「1千ドル(10万円)、数時間」で個人のDNAシークエンス情報が得られる装置が実用化を迎え、2、3年後くらいには、安定的に利用できるようになる見込みです。さらに「100ドル、1時間以内」というシークエンサーの実現も間近かといわれています。こうした超安価・高速・高精度のシークエンサーの普及に伴って、自分のDNAを調べる臨床シークエンスに基づいて病気の診断、治療の方針や薬の種類・量などを決める個別化医療は急速に広がり、誰もが自分のDNAシークエンスを持つことができる時代も、それほど遠くないと思っています。そして、パーソナルゲノム時代が本格化すれば、膨大なゲノムデータが生まれ、生命システムの理解にも新たなブレークスルーがもたらされることでしょう。
そのさきがけともいえる成果は、すでに生まれています。それが「骨髄異形成症候群(MDS)の原因遺伝子の発見」です。骨髄異形成症候群は、骨髄で正常な血液がつくれなくなることにより、急性骨髄性白血病へと移行していくケースが多い病気です。これまでその原因は不明でしたが、東京大学医学部附属病院キャンサーボードの小川誠司らの研究チームは、東京大学医科学研究所ヒトゲノム解析センターのスーパーコンピュータをフルに用い、次世代シークエンサーを使って、検体のエクソン(RNAスプライシング反応で残る部位)の解析を徹底的に行うことにより、RNAスプライシングの経路の4つの遺伝子に現れる変異がその原因であることを明らかにしました。初めてこの病気の原因遺伝子を発見したこととともに、RNAスプライシングの異常ががんの発症に関わることを世界で初めて示した研究成果であり、がん研究の歴史に残る成果ともいわれています。これは、まさにシークエンス技術とスーパーコンピュータ、統計的数理解析チームが一緒になってなし遂げた成果のひとつといえます。現在、私たちが取り組んでいるHPCI戦略プログラム(戦略分野1)の「大規模生命データ解析」においても、こうしたブレークスルーの成功例を生み出していきたいと考えています。
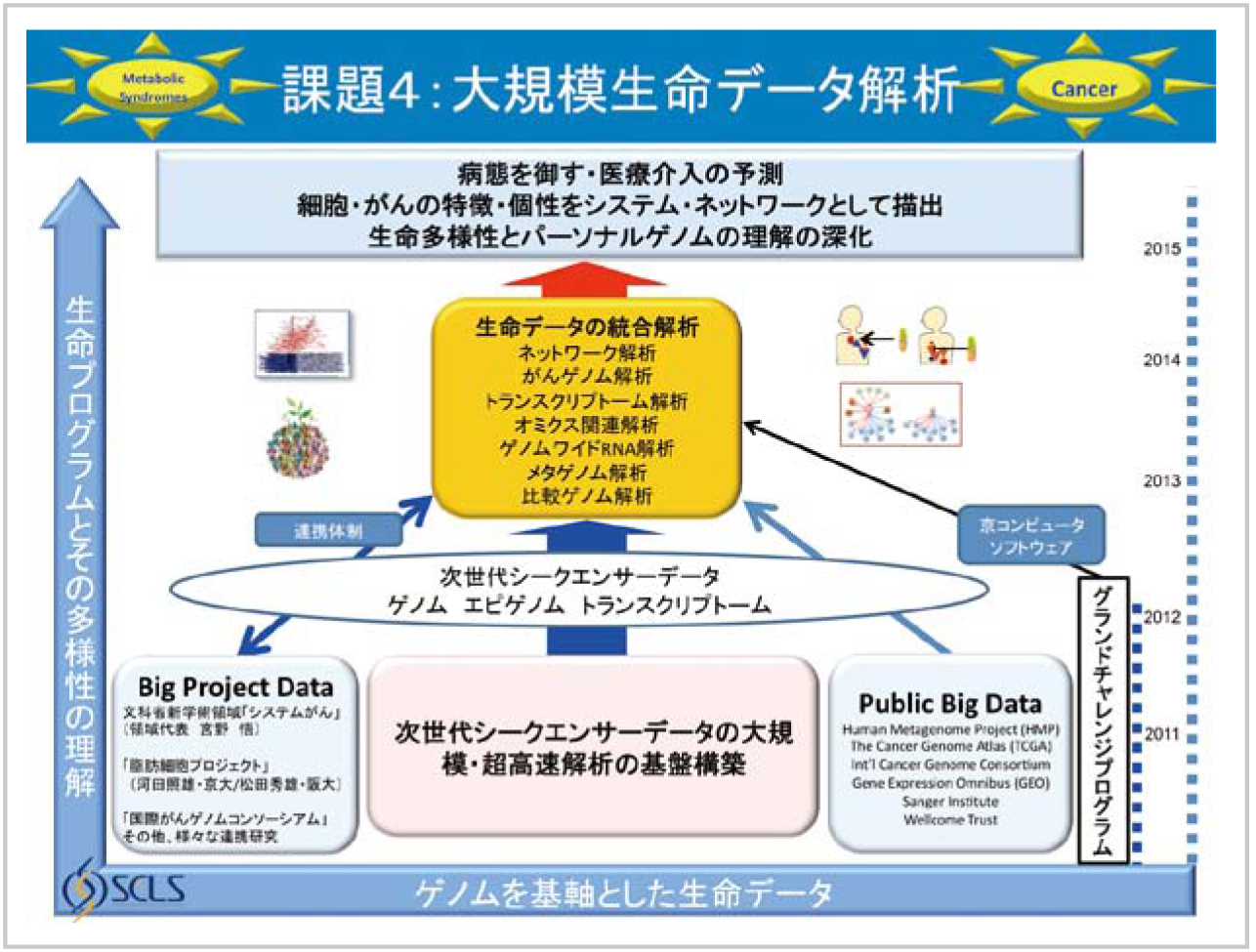
課題4の研究推進計画
●課題4が取り組む研究内容
課題4「大規模生命データ解析」の大きな目的は、生命プログラムとその多様性の理解です。この目的を達成するためにまず必要となるのが、次世代シークエンサーデータの大量高速処理です。そのために2011 ~ 12年度は、「京」に最適化した次世代シークエンサーデータの大規模・超高速解析の基盤構築に力を注いでいます。この解析システムでは、毎時1,000万リードの性能を達成することにより、大量のゲノム配列情報の高速解析を実現させ、より難度の高い、微小な類似性を感知できる世界最深度の高速配列探索を行い、世界に対して圧倒的な優位性を確保する体制づくりをめざしています。
解析の基盤となるデータに関しては、パブリックな公開データベースに加えて、がんのTCGAプロジェクト、メタゲノムプロジェクト、さらに課題4の研究者が参加する国際がんゲノムコンソーシアム、新学術領域・システムがん、脂肪細胞プロジェクトなど、さまざまな研究プロジェクトと連携し、実験による検証もこうしたプロジェクトのなかで行うことになります。
具体的な研究内容は、「がんのリネッジと多様性の理解」、「パーソナルゲノム理解の深化」、「個別化医療介入予測」、「細胞分化ネットワーク」、「薬剤応答ネットワーク」、「パーソナルがんネットワーク」など幅広い研究内容が展開されますが、その到達点として3つを挙げることができます。1つ目は、「生命の多様性とパーソナルゲノムの理解の深化」です。なかでもメタゲノムとがんの多様性に重きを置き、さらにパーソナルゲノムの理解を進化させていくことで、生命の多様性の理解を深めていけるだろうと考えています。2つ目は、「細胞やがんの特徴・個性をシステムやネットワークとして抽出」していくことです。そして、3つ目は、医療介入の予測につなげていくことによって「病態を御す」ということです。これらの3つをめざして、研究を進めていこうとしています。
ライフサイエンス分野におけるシミュレーション研究が盛んに行われていますが、現在は、まだ大腸菌ひとつとっても、完全にシミュレーションできるまで生命システムが明らかにされているとはいえません。ましてヒトの場合は、理解は進んでいるものの、数理モデルを構築してシミュレーションすることにより、新たなバイオロジカルな知見を導き出したり、誰にでも効く薬を開発するというのは、とても難しいのが実情ではないでしょうか。課題4では、まずデータからスタートして、データドリブンのサイエンスを行っていこうと考えています。データを統合的に解析し、そこから見えてくるものを明らかにすることによって、個人個人の病気の原因の糸口を探し出していくための手法を開発していく、それが私たちの取り組み方です。
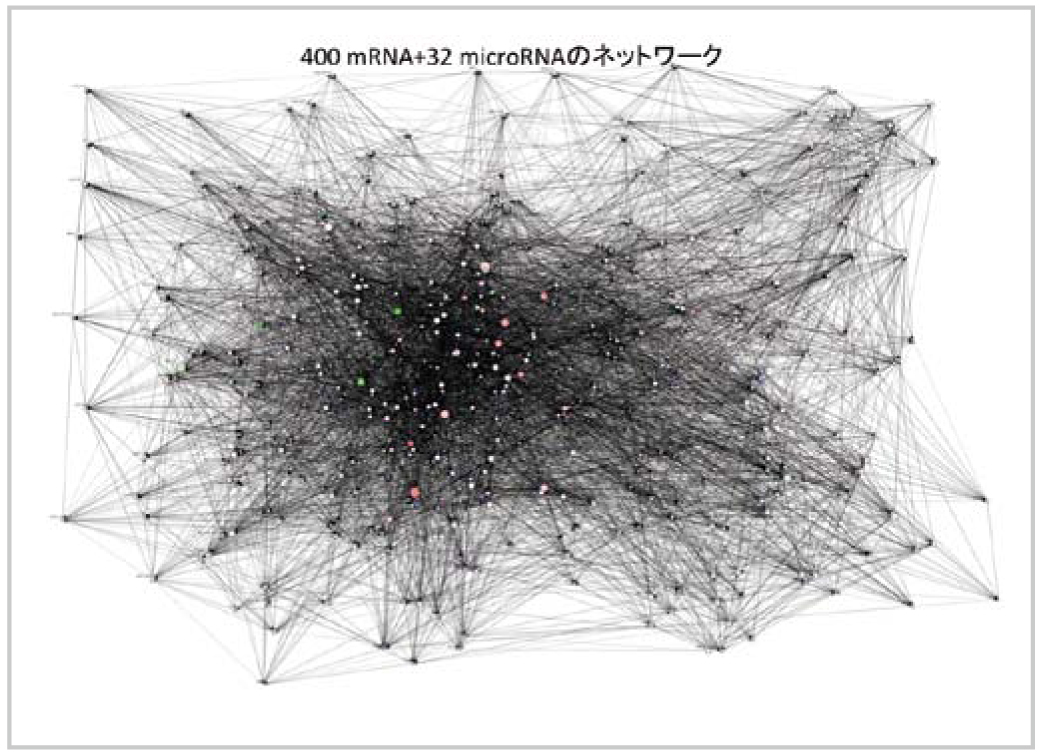
肺線がんのマイクロRNA/mRNA遺伝子ネットワーク。遺伝子間の因果関係をネットワークでとらえた全体図。
(名古屋大学医学研究科・高橋 隆教授提供)
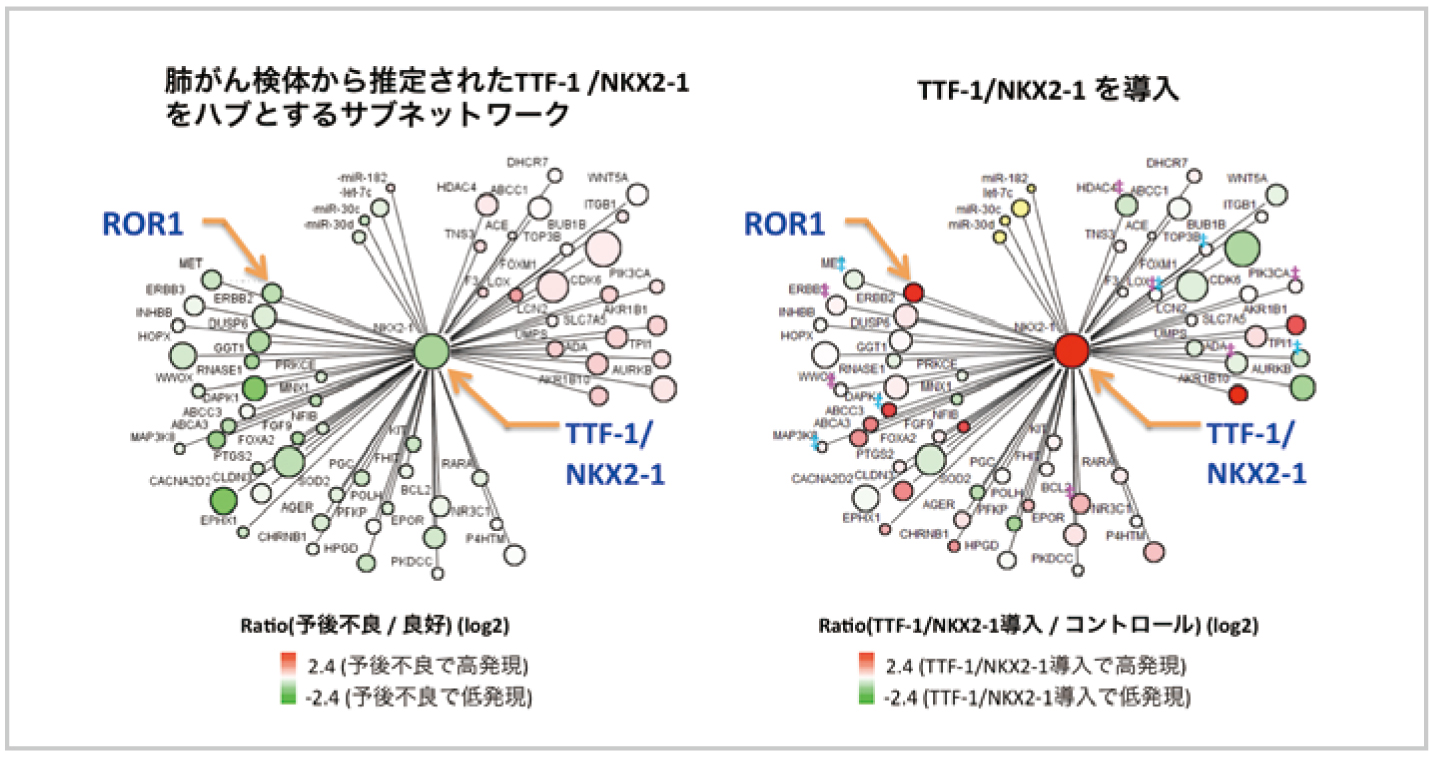
TTF-1/NKX2-1遺伝子を導入すると有意な関係を示すまわりのネットワークの発現が大きく変化し、肺線がんが生き残るかどうかのスイッチが変わったことが明らかになった。
(名古屋大学医学研究科・高橋 隆教授提供)
BioSupercomputing Newsletter Vol.8
- SPECIAL INTERVIEW
- 革新的なアプローチでライフサイエンス分野の未来を切り拓いてきたグランドチャレンジ
理化学研究所 次世代計算科学研究開発プログラム
プログラムディレクター 茅 幸二 - ライフサイエンス分野の研究開発に革新をもたらした画期的なプロジェクト
理化学研究所 次世代計算科学研究開発プログラム
副プログラムディレクター 姫野 龍太郎
- 研究報告
- マルチスケール・マルチフィジックス心臓シミュレータUT-Heart
東京大学新領域創成科学研究科
久田 俊明、杉浦 清了、鷲尾 巧、岡田 純一、高橋 彰仁
(臓器全身ケールWG) - 膵臓β細胞内インスリン顆粒動態シミュレーション・モデル
神戸大学大学院システム情報学研究科 玉置 久(細胞スケールWG) - 京による全脳シミュレーションへの道のり
理化学研究所 脳科学総合研究センター ユーリッヒ研究センター
神経科学・医療研究院(INM-6)
アーヘン工科大学医学部 マーカス・ディースマン(脳神経系WG) - 大規模並列用MDコアプログラムの開発
理化学研究所 次世代計算科学研究開発プログラム 大野 洋介(開発・高度化T)
- SPECIAL INTERVIEW
- 循環器系および筋骨格系・脳神経系における階層統合シミュレーションの実現をめざす
東京大学大学院工学系研究科機械工学専攻 教授 高木 周(課題3 代表) - 最先端シークエンサーによる大規模データを「京」で解析し生命プログラムとその多様性の理解を進める
東京大学医科学研究所ヒトゲノム解析センター 教授 宮野 悟(課題4代表)
- 報告
- 「4th Biosupercomputing Symposium」の開催報告
理化学研究所 次世代計算科学研究開発プログラム 田村 栄悦 - 京互換機:SCLS計算機システムの導入
理化学研究所 HPCI計算生命科学推進プログラム チーム員 木戸 善之
