京による全脳シミュレーションへの道のり

理化学研究所 脳科学総合研究センター
ユーリッヒ研究センター 神経科学・医療研究院(INM-6)、アーヘン工科大学医学部
マーカス・ディースマン
(脳神経系WG)
脳神経系研究開発チーム(BNT)が、京の計算能力を計算論的神経科学の分野において利用可能となるようにソフトウェア開発を進めてきた経緯をここで紹介します。本レポートの拡張版は[1]で見ることができます。
ヒトの脳は約1011個の神経細胞で構成されており、各々が10000個の別の神経細胞と結合しています。計算論的神経科学では、神経回路のダイナミクスを解明するためのボトムアップアプローチとして、しばしば、神経細胞およびそれらの相互関係をモデルとして記述することから始めます[2]。NESTシミュレーター[3]はこうしたアプローチに向いたツールです。神経細胞は、微分方程式からなる小さなシステムとして記述され、δインパルス[4, 5]によって相互に情報交換することで、自然な規模と複雑さをもつ回路を構成します。
トップダウンアプローチでは、特定の脳機能を大まかに記述することから始め、この機能がどのように神経細胞レベルで実行されているかを調べます(例:[6])。脳の機能的回路は、通常多くの脳領野により成り立ちます。しかし、これまでのシミュレーションでは、主に計算機のメモリの必要性から[7]、局所回路(例:[8, 9])またはスパイクベースでない巨視的モデルに制限されていました。京のようなスーパーコンピュータを用いると、脳のようにマルチスケールな結合をもつ巨大システムのシミュレーションも可能となります。
第1回BNT会議(2008年11月)の時点では、NEST(第2世代カーネル、2g)は、105個の神経細胞からなる回路シミュレーションを1024プロセッサにより実施していました。第3回会議(2009年10月)では、ユーリッヒ研究所のJUGENEコンピュータを用いることで、106個の神経細胞によるシミュレーションが可能になりました。これを受けて私たちは、2010年の2月までに、京の上で動作させる回路における神経細胞数を106から108個以上へと拡大することを、マイルストーンとして設定しました。NESTは2010年11月に京のプロトタイプで、2011年5月に試験システムで、そして同年9月には京で、コンパイルすることに成功しました。
3gカーネルでは、神経細胞の局所結合が疎であるという性質を考慮したデータ構造となり、あわせて、神経細胞の非局所結合に関する情報を効率的に保存することができるようになりました[7]。2012年3月の第8回BNT会議では、次のマイルストーンを視野に入れることが示され、5月[10]には、京の利用ノード数として12288に到達しました。近々公開されるNEST 2.2は、このカーネルが基礎となっています。
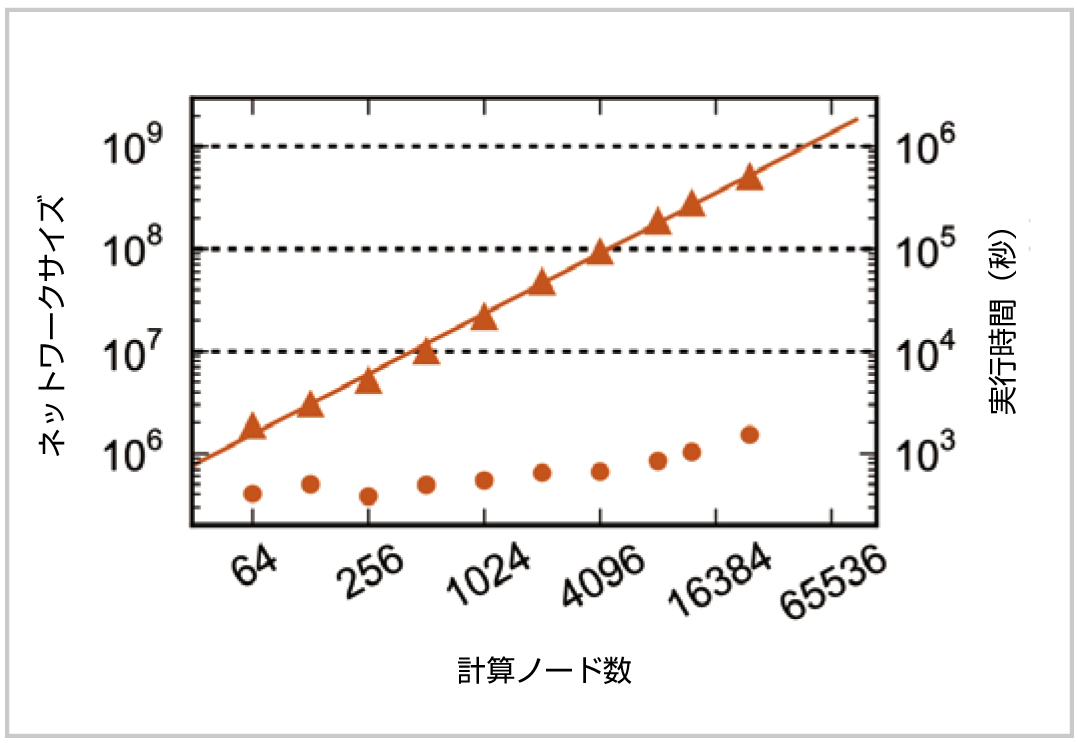
神経細胞間の結合(シナプス)には、異なるタイプのダイナミクスと可塑性を持つものがあります[11,12]。しかし、一つの神経細胞が、各計算機コア上で、シナプスを持たない、または一個のシナプスだけを持つ場合に制限すると、このシナプス構造の非一様性は無視でき、単一種のシナプスのみからなるようなものと等価に扱うことができます。2012年9月の第9回会議で発表された4gカーネルは、この特性を利用しています。図は、横軸のノード数を用いることでシミュレーション可能な最大回路サイズ(三角)およびその実行時間(点)を示しています。4gカーネルにより、京上で109個の神経細胞からなる回路のシミュレーションが可能となることが分かります。NESTのユーザインタフェースは同じままで、カーネルの一般性および機能性はどちらも損なわれていません[13,14]。
私たちは、神経科学者が、脳の解剖学およびダイナミクスに関するデータをモデルとして統合することで脳機能に関する洞察を得る際に、シミュレーション技術が役立つようになったと考えています。
今後は、4gで新たに開発された技術をNESTの公開版として実装し、さらに、現在準備段階にあるエクサ規模の計算機のためのソフトウェアの開発に取り組みます。
www.csn.fz-juelich.de
www.nest-initiative.org
謝辞
本研究の一部は、理化学研究所 計算科学研究機構での京のアーリーアクセス、文部科学省の次世代スーパーコンピュータ戦略プログラム、ヘルムホルツ協会、HASBおよびポートフォリオテーマSMHB、JARA、並びにEU助成金269921(BrainScaleS)によるものです。
|
【参考文献】
[1] Diesmann (2012) p83-85 In Proc 4th BioSupercomputing Symposium,Tokyo
[2] Lansner and Diesmann (2012) Chap 10 In Computational Systems Biology, Le Novere ed., Springer
[3] Gewaltig and Diesmann (2007) Scholarpedia 2(4):1430
[4] Morrison et al. (2008) Biol Cybern 98:459-78
[5] Hanuschkin et al. (2010) Front Neuroinform 4:113
[6] Potjans et al. (2011) PLoS Comput Biol 7(5): e1001133
[7] Kunkel et al. (2012) Front Neuroinform 5:35
[8] Potjans et al. (2012) Cerebral Cortex doi:10.1093/cercor/bhs358
[9] Wagatsuma et al. (2011) Front Comput Neurosci 5:31
[10] Helias et al. (2012) Front.Neuroinform 6:26
[11] Potjans et al. (2010) Front Comput Neurosci 4:141
[12] Pfeil et al. (2012) Front Neuromorph Eng 6:90
[13] Eppler et al. (2009) Front Neuroinform 2:12
[14] Gewaltig et al. (2012) Chap 18 In Computational Systems Biology, Le Novere ed., Springer
BioSupercomputing Newsletter Vol.8
- SPECIAL INTERVIEW
- 革新的なアプローチでライフサイエンス分野の未来を切り拓いてきたグランドチャレンジ
理化学研究所 次世代計算科学研究開発プログラム
プログラムディレクター 茅 幸二 - ライフサイエンス分野の研究開発に革新をもたらした画期的なプロジェクト
理化学研究所 次世代計算科学研究開発プログラム
副プログラムディレクター 姫野 龍太郎
- 研究報告
- マルチスケール・マルチフィジックス心臓シミュレータUT-Heart
東京大学新領域創成科学研究科
久田 俊明、杉浦 清了、鷲尾 巧、岡田 純一、高橋 彰仁
(臓器全身ケールWG) - 膵臓β細胞内インスリン顆粒動態シミュレーション・モデル
神戸大学大学院システム情報学研究科 玉置 久(細胞スケールWG) - 京による全脳シミュレーションへの道のり
理化学研究所 脳科学総合研究センター ユーリッヒ研究センター
神経科学・医療研究院(INM-6)
アーヘン工科大学医学部 マーカス・ディースマン(脳神経系WG) - 大規模並列用MDコアプログラムの開発
理化学研究所 次世代計算科学研究開発プログラム 大野 洋介(開発・高度化T)
- SPECIAL INTERVIEW
- 循環器系および筋骨格系・脳神経系における階層統合シミュレーションの実現をめざす
東京大学大学院工学系研究科機械工学専攻 教授 高木 周(課題3 代表) - 最先端シークエンサーによる大規模データを「京」で解析し生命プログラムとその多様性の理解を進める
東京大学医科学研究所ヒトゲノム解析センター 教授 宮野 悟(課題4代表)
- 報告
- 「4th Biosupercomputing Symposium」の開催報告
理化学研究所 次世代計算科学研究開発プログラム 田村 栄悦 - 京互換機:SCLS計算機システムの導入
理化学研究所 HPCI計算生命科学推進プログラム チーム員 木戸 善之