
次世代DNAシークエンサデータの超高速解析
HPCI戦略プログラム 分野1 予測する生命科学・医療及び創薬基盤
東京工業大学大学院情報理工学研究科
(左から)秋山 泰、石田 貴士、角田 将典、鈴木 脩司 (分野1-課題4)




次世代DNAシークエンサは、飛躍的な技術革新により1回の走査で約1兆文字ものDNA配列読み取りを可能にしている。いわゆる旧型シークエンサの時代でも「1ドル当たりで読み取れる文字数」は約19ヶ月で2倍という驚異的な伸び率であったが、次世代シークエンサの登場以降は約5ヶ月で2倍の伸び率と言われ、また世界的に圧倒的な台数が普及しつつある。現在では読み取られた配列の意味解釈の計算が最大のボトルネックである。遺伝子ネットワークの推定のような総合的な高度計算にスパコンが必要な事は周知の事実であるが、そればかりか配列断片群に対する基本的処理にすら大規模計算が必要となった。
ヒトの遺伝子のわずかな変異などの研究では、既知配列との完全一致または強く類似した文字列の探索に適した高速アルゴリズムを使うことができ、大型のPCクラスタを用いて何とか対処されてきた。しかし一方、ヒトの腸内等の微生物のゲノム断片を網羅的に調べあげる研究や、深刻な配列の変異を扱う研究では、遠縁あるいは大きく変異した配列間での配列相同性を検知しなければマッピング(既知配列との対応付け)が実行できない。これには桁違いに大きな計算が必要となり、PCクラスタ等では歯が立たない。
ヒトの健康は体内に共生する多数の微生物のバランスの取れた働きにより維持されている。またそのバランスには幾つかの局所安定解があると考えられている。米国のHumanMicrobiomeProject(HMP)では約500名、欧州のMetaHITプロジェクトでは約300名のヒトの口腔内、消化管内、皮膚、泌尿生殖系等の環境での微生物群のゲノム断片の網羅的な読み取りが実行されているが、このような貴重なデータを分析する際に、文字列の強い類似性を仮定した感度の悪い手法だけでは、解釈不能として読み捨てられる断片配列の比率が大きく増加してしまう。
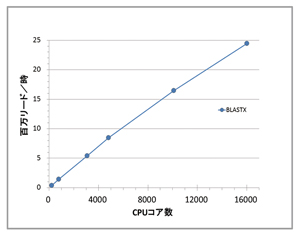
我々は、ゲノム解析の専門家である黒川顕教授(東工大)の協力の下に、まず既存のBLASTXソフトウェアの超並列実行環境の構築を行った。東工大TSUBAME2.0上では16000コア超までのスケーラビリティを確認した(図1)。各計算ノードへのデータベースの初期配送では、ネットワークのバンド幅を活かした二分木転送を用いた。我々はDNA断片配列を可能な6通りの読み枠でアミノ酸配列に変換してから、既知アミノ酸配列との柔軟な比較を行う。DNA配列レベルでは大きく変異していても、アミノ酸レベルでは残基が一致する、あるいは似た物理化学的性質を持つ残基に変異していることが多いため、文字間の距離行列を用いて比較する柔軟な手法を用いることにより、同じ機能をもつ遠縁の遺伝子を探す検出感度が高まるためである。
次に、我々はアルゴリズム上の大幅な高速化を行い、BLASTXを改良したGHOSTXと称するソフトウェアを作成した[1,2]。GHOSTXでは、メモリに格納された区分データベースをSuffixArrayで表現し、またクエリ配列側もSuffixArrayに変換し、この両者を効率良く比較することで、同じ部分文字列の一致を多対多で一気に解決できる(図2)。またBLASTXでは固定長の文字列比較しか行わないのに対し、GHOSTXでは一致スコアを満たすまで可変長で文字列を伸ばせるため、計算コストあたりの感度が向上する。BLASTXと同等の感度が必要な場合でも速度は約20倍。実用上支障がない程度に感度を下げると100倍超の高速化が達成できた。高速だが感度が粗すぎて利用できなかったBLAT法より感度は大幅に良く、速度も2〜3倍速いという結果も得ている。我々は「京」の上にGHOSTXアルゴリズムを実装し、OpenMPによるスレッド並列化と、MPIによるノード間のデータ並列性を組み合わせて、“GHOST-MP”と呼称する超並列解析システムを構築した。
相同性検索の主要部では、「京」で1536ノード(12288コア)までの良好なスケーラビリティを実測した。断片配列1本ごとの実行時間は千差万別だが、負荷分散機構を有するため、数万ノードまでのスケーラビリティが期待できる。GHOST-MPでは、読み込むデータベースとクエリ配列群の長さ同士の積のオーダーの計算を行うので、I/Oに終始する処理ではないものの、注意深いI/O設計は必要である。「京」ではZ軸方向の通信でI/Oノードとの入出力転送が実施されるため、代表ノードによるデータ入力とブロードキャストの機構を調整してI/O負荷のさらなる分散と低減を図り、数万ノード級までの高効率実行を目指す。
【参考文献】
[1] 鈴木, 石田, 秋山, 情処研報 2010-BIO-23(21), 1-6 (2010).
[2] 鈴木, 石田, 秋山, 情処研報 2011-BIO-25(32), 1-8 (2011).
 |
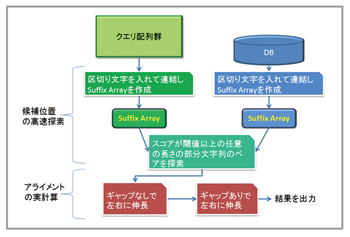 |
図1:BLASTXベースのシステムでの並列性能 |
図2:GHOSTXアルゴリズムにおける処理の流れ |
BioSupercomputing Newsletter Vol.5
- SPECIAL INTERVIEW
- “予測する生物学”をめざすバイオスーパーコンピューティングの挑戦はいよいよ世界一の「京」で成果を出すフェーズに入った
理化学研究所 次世代計算科学研究開発プログラム 副プログラムディレクター 姫野 龍太郎 - 高性能計算機資源および開発アプリケーションの産業利用促進を図るために何をすべきか
計算科学振興財団 チーフコーディネーター 福田 正大
都市活力研究所 主席研究員 バイオグリッドセンター関西 理事・事務局長 志水 隆一
- 研究報告
- QM/MM 自由エネルギー法による酵素反応分子機構の解析
京都大学大学院理学研究科 林 重彦(分子スケールWG) - アクチン細胞骨格の計算メカノバイオロジー
京都大学再生医科学研究所 井上 康博(細胞スケールWG) - 血栓シミュレーションに向けた血流解析手法の開発
東京大学工学系研究科 伊井 仁志(臓器全身スケールWG) - 生命体シミュレーションのためのデータ同化技術の開発
統計数理研究所 樋口 知之(データ解析融合WG)
- SPECIAL INTERVIEW
- 複雑な生命現象の理解と予測に向けて計算生命科学の明日を拓く
理化学研究所 HPCI計算生命科学推進プログラム プログラムディレクター 柳田 敏雄
理化学研究所 HPCI計算生命科学推進プログラム 副プログラムディレクター 木寺 詔紀
理化学研究所 HPCI計算生命科学推進プログラム 副プログラムディレクター 江口 至洋
- 研究報告
- 創薬応用シミュレーション
東京大学先端科学技術研究センター 藤谷 秀章(分野1-課題2) - 次世代DNAシークエンサデータの超高速解析
東京工業大学大学院情報理工学研究科
秋山 泰 / 石田貴士 / 角田将典 / 鈴木脩司(分野1-課題4)
- 報告
- バイオスーパーコンピューティングサマースクール2011
理化学研究所 次世代計算科学研究開発プログラム
石峯 康浩(臓器全身スケールWG)
統計数理研究所 データ同化研究開発センター
斎藤 正也(データ解析融合WG)
新潟国際情報大学
近山 英輔(細胞スケールWG)
東海大学医学部内科学系循環器内科
七澤 洋平(細胞スケール/臓器全身スケールWG)
理化学研究所 次世代計算科学研究開発プログラム
半田 高史(脳神経系WG)
理化学研究所 次世代計算科学研究開発プログラム
舛本 現(開発・高度化T)
理化学研究所 次世代計算科学研究開発プログラム
森次 圭(分子スケールWG) - 「次世代生命体統合シミュレーションソフトウェアの開発(ISLiM)」開発アプリケーション紹介ページ、オープン
次世代計算科学研究開発プログラム 次世代生命体統合シミュレーション研究推進グループ
