(Updated on 2016/1/28)
理化学研究所生命体基盤ソフトウェア・高度化チーム
大野洋介
高並列に特化した高速な古典分子動力学計算
古典分子力場、Lennard-Jonesモデル、クーロン力
近距離直接和と遠距離FFM/PME
空間分割(MPI並列)、ループ分割(スレッド並列)
C/C++, MPI, OpenMP
ソース・コードをISLiMダウンロードサイトから公開済み。
- 98万原子系
- RICC 8000 コア並列
- 1000ステップ5.1秒 通信時間4.5秒
- メモリ容量 1.2 TB、ディスク容量 0.1 TB
- 百万原子系でサブミリ秒のシミュレーション
- 積分時間約サブミリ秒
- 反復回数109回以上
- メモリ容量 10 TB、ディスク容量 100 TB
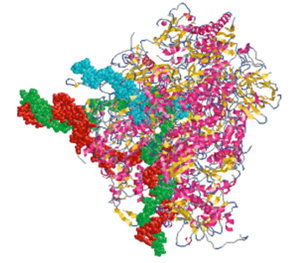
超並列大規模分子動力学計算の例
- 64万コア以上の超並列MD計算が可能となる。
- 生体分子の運動の長時間計算が可能となる。
100万原子系のサブミリ秒シミュレーションを目指す。 - 生命システムの原子レベルでの構成原理を理解すると共に創薬に必要なレベルの計算精度を実現する。
- やさしいcppmd (超並列高速MD計算コア)ソフトの紹介と利用方法の詳細 -> リンク
理化学研究所生命体基盤ソフトウェア・高度化チーム
小野謙二
大規模シミュレーションの結果を分散並列して可視化する.リモート可視化,スタンドアロン可視化,インタラクティブ可視化,バッチ可視化などさまざまな利用形態に対応する並列可視化アプリケーション.クライアントはマルチプラットホームで動作する
領域分割によるMPI並列
複数のレプリカの並列実行
C++, MPI, libxml2, zlib
- ソース・コードをISLiMダウンロードサイトから公開済み
- 到達並列数100コア
- データ格子サイズ:2450 x 1330 x 8435, 約100GB
- 数十万コアを用いた並列実行
- メモリ容量 100TB、ディスク容量 1 PB
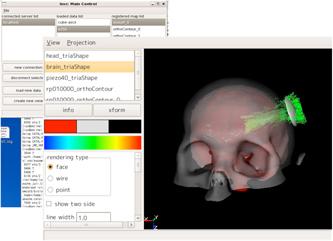
HIFU(低侵襲治療シミュレーション)データの可視化の例
- PCでは可視化不可能な大規模データの可視化
- サーバで可視化した結果をインタラクティブに観察するリモート可視化と動画作成を目的としたバッチ可視化
- プラグインによる可視化機能の拡張
理化学研究所生命体基盤ソフトウェア・高度化チーム
小野謙二
連続体力学シミュレーションコードの開発・運用を促進する統合開発環境。離散化手法は直交格子系および非直交格子系の双方を対象とする。各ハードウェア用自動最適化効果も含む。
差分法、有限要素法、有限体積法
反復法による疎行列連立方程式求解
空間分割
C++, Fortran90, MPI, OpenMP, zlib, libxml2
ソース・コードをISLiMダウンロードサイトから公開済み
- 三次元熱伝導方程式のベンチマークテスト
- ボクセル数8192x2048x2048
- RICC 8192コア
- メモリ容量 4TB、ディスク容量 1 TB
物理シミュレーション全般
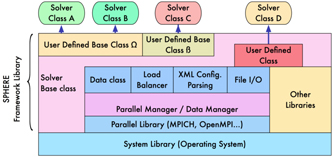
SPHEREフレームワークライブラリ
- SPHEREは、物理シミュレータ構築に利用するアプリケーションミドルウェアで、できることは構築したシミュレータに依存。
東京大学工学系研究科教授 船津公人
創薬スクリーニングのためのバーチャルライブラリには、規模と多様性が求められる一方、バーチャルであるがゆえに、高スコアのリード化合物群を絞り込んでも、それらの合成検討に多大なコストが強いられるという問題が残る。また、化合物創生を原子種の組み合わせと、各原子の取りうる結合次数による論理操作だけに頼ると、合成不可能な不安定化合物を多く含む出力を得てしまう。これらの問題を 回避しつつ、従来にない大規模なバーチャルライブラリを提供する。
反応データベースから抽出された反応前後の反応部位構造変化トランスフォームを活用し、標的構造に対する前駆体構造を反応スキームとして提示できるシステムを連続運用
Fortran
未公開
- 42万の種化合物群から、1段階で630万の仮想化合物、1200万の仮想反応スキームを得た。
- 1段目の出力構造を再度入力構造として、1億件の化合物出力を得た。
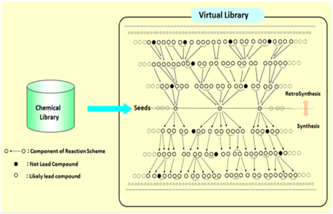
バーチャルライブラリの構成
- 合成ルートが付与された大規模バーチャルライブラリの実現
・入手可能な化合物に至る合成経路の提示
・複数の合成経路のコスト評価 - 出力構造の新規性確保
1段目の出力構造630万件と、1500万分子を含む市販試薬ライブラリとの対比では、0.004%しか重複しなかった。 - 存在しえない構造の僅少化。