
大規模並列用MDコアプログラムの開発

理化学研究所 次世代計算科学研究開発プログラム
大野 洋介
(開発・高度化T)
我々のチームでは、「京」用に高速化されたコアプログラム・ライブラリの提供および「京」でのアプリケーション高速化技術の習得・蓄積を目的として、大規模並列用MDコアプログラムを開発しています。
分子動力学(Molecular Dynamics, MD)シミュレーションは分子を構成する原子に働く力と運動を計算することで分子の運動・構造変化等を再現する手法の一つです。
生命科学においては、生命現象の基盤となるタンパク質のような生体分子の働きを解明するのに利用されています。タンパク質等の生体分子のシミュレーションでは、原子に働く力として、共有結合による力、ファンデルワールス力、クーロン力を扱います。共有結合は調和振動子のような単純な古典力場で代用し結合が連続したせいぜい4個の原子までしか扱わないので計算量は共有結合する原子の個数にほぼ比例しますが、任意の原子間に働くファンデルワールス力とクーロン力では全ての原子の組み合わせを扱うので計算量は原子の個数の二乗に比例し、原子の個数が多い場合はファンデルワールス力とクーロン力の計算が処理時間の大半を占めるようになります。高速化の手法としてもっと単純なのは遠距離の原子間の力を無視するカットオフ計算です。ファンデルワールス力は距離の7乗、13乗に反比例して小さくなるので、1.4nm以遠を無視してもほとんど計算精度に影響しません。クーロン力は距離の2乗に反比例して小さくなるだけなので、単純なカットオフ計算では計算精度に影響します。そのため、遠距離のクーロン力を高速に計算する手法としてFFTを利用するPME(Particle Mesh Ewald)法が主流となっています。しかし、FFTは広い範囲の通信が必要で並列度が高くなると通信時間の増加が問題になるため、高速多重局展開法(FMM)等のFFTを使わないの計算手法を導入するといった対策を検討しています。
カットオフ計算による大規模テスト計算として、“分子の混み合い”のタンパク質動態への影響を調べるためのシミュレーションを行ないました。細胞内環境では通常の実験より高濃度でタンパク質が存在しており、タンパク質の混み合いが構造や相互作用に影響を与えていることが実験的にも示されています。タンパク質の混み合いを再現するには多数のタンパク質を含んだシミュレーションを行なう必要となるため、大規模計算が可能な「京」の活躍が期待されます。テスト計算では、水中と卵白のタンパク質ovalbumin(原子数6000弱)が30%含まれる環境で、TTHA1718 という1000原子程度のタンパク質の構造を比較しました。図1はTTHAを構成するアミノ酸の位置の揺らぎの大きさを示しています。計算時間が短く実験を再現するには至っていませんが、水中の場合(赤線 vit1、青線 vit2)とovalbuminを含む場合(緑線viv)でTTHAの動きが異なることは確認できました。図2はTTHAがovalbuminに囲まれている様子を可視化したものです。
「京」の性能評価(注)での実行では、1億8000万原子、カットオフ2.8nmの計算で、効率30%以上、実効計算速度1.3PFlops という性能が得られました。カットオフ計算部分は効率40%以上を達成しています。
現時点でカットオフ計算とPME 法・FMMが実装済みです。PME 法・FMM等は詳細なアルゴリズムの変更も含めた最適化を行なっています。「京」のメッシュ/トーラスネットワークの構造に合わせた通信方式を実装していますが、ノードあたりの原子数が少ない場合は計算時間より通信時間の方が長くなるため、通信時間を短縮する改良も継続しています。
本研究は高度化チーム小山洋、舛本現、長谷川亜樹、森本元太郎、沖本憲明、平野秀典、生命システム研究センター宮下尚之との共同研究です。
本研究の結果の一部は、RIKEN Integrated Cluster of Clusters(RICC)システムを利用して得られました。
(注) 理化学研究所次世代スーパーコンピュータ開発実施本部が実施している次世代スーパーコンピュータの開発・整備における京速コンピュータ「京」の性能評価。
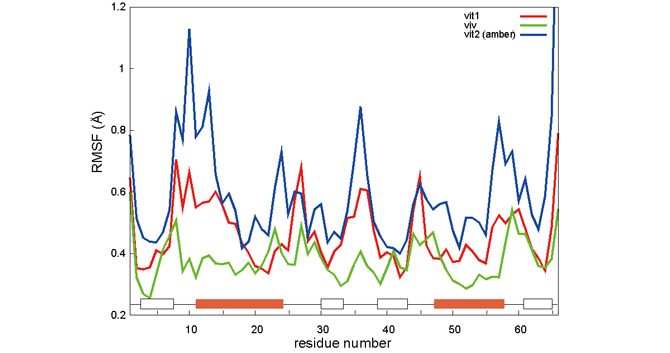
図1:TTHAの構造の揺らぎの大きさ 赤・青は水中、緑はovalbuminを含む場合。横軸はアミノ酸の番号、縦軸は揺らぎの大きさ。
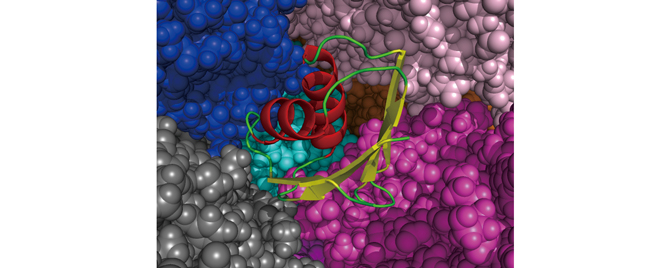
図2:ovalbuminに囲まれたTTHA中央のらせんや矢印で表現されているのがTTHA、周囲の球はovalbuminの原子で、一色が一個のovalbuminを構成。
BioSupercomputing Newsletter Vol.6
- SPECIAL INTERVIEW
- 新しい流体構造連成解析手法(ZZ-EFSI)の開発によっていち早く高い演算性能を達成
東京大学大学院工学系研究科 特任准教授 杉山 和靖 - 開発・高度化チームに聞く「京」の実力と高い性能を引き出すために続くチューニングの取り組み
理化学研究所 生命システム研究センター 生命モデリングコア計算分子設計研究グループ
グループディレクター 泰地 真弘人
理化学研究所 次世代計算科学研究開発プログラム 次世代生命体統合シミュレーション研究推進グループ
生命体基盤ソフトウェア開発・高度化チーム 上級研究員 大野 洋介
理化学研究所 HPCI計算生命科学推進プログラム 高度化推進グループ
高度化推進チーム 上級研究員 小山 洋
理化学研究所 次世代計算科学研究開発プログラム 次世代生命体統合シミュレーション研究推進グループ
生命体基盤ソフトウェア開発・高度化チーム 研究員 舛本 現
理化学研究所 次世代計算科学研究開発プログラム 次世代生命体統合シミュレーション研究推進グループ
生命体基盤ソフトウェア開発・高度化チーム リサーチアソシエイト 長谷川 亜樹
- 研究報告
- 全原子分子動力学シミュレーションによる多剤排出トランスポーターAcrBの機能解析
横浜市立大学大学院生命ナノシステム科学研究科 山根 努 / 池口 満徳( 分子スケールWG) - ヒト循環器系のマルチスケールモデリング
理化学研究所 次世代計算科学研究開発プログラム 梁 夫友( 臓器全身スケールWG) - 初期サッカード視覚運動系のスパイキングニューロンレベルでのモデル化
京都大学 ジャン・モーレン
奈良先端科学技術大学院大学 柴田 智広
沖縄科学技術大学院大学 銅谷 賢治
(脳神経系WG) - 大規模並列用MDコアプログラムの開発
理化学研究所 次世代計算科学研究開発プログラム 大野 洋介(開発・高度化T)
- SPECIAL INTERVIEW
- 複雑な生命現象の理解と予測に向けて計算生命科学の明日を拓く
理化学研究所 HPCI計算生命科学推進プログラム プログラムディレクター 柳田 敏雄
理化学研究所 HPCI計算生命科学推進プログラム 副プログラムディレクター 木寺 詔紀
理化学研究所 HPCI計算生命科学推進プログラム 副プログラムディレクター 江口 至洋
- 研究報告
- 全原子モデルにもとづくヌクレオソームポジション変化の自由エネルギープロファイル計算
日本原子力研究開発機構 量子ビーム応用研究部門
河野 秀俊 / 石田 恒 / 米谷 佳晃 / 池部 仁善
(分野1-課題1) - 骨格筋の活動の推定と脊髄反射の神経モデル
東京大学情報理工学系研究科 中村 仁彦(分野1-課題3)
- 報告
- ISLiM 成果報告会2011
理化学研究所 次世代計算科学研究開発プログラム 田村 栄悦 - 高等学校で行った計算生命科学の授業
理化学研究所 HPCI計算生命科学推進プログラム 鎌田 知佐 / 藤原 康広 / 江口 至洋
