
全原子分子動力学シミュレーションによる
多剤排出トランスポーターAcrBの機能解析


横浜市立大学大学院生命ナノシステム科学研究科
山根 努 (写真左)
池口 満徳 (写真右)
(分子スケールWG)
近年、医療現場での薬物療法において、病原菌やがん細胞などに対して薬が効かなくなる現象である薬剤耐性化が大きな社会問題となっています。このような薬剤耐性化のメカニズムはいくつか知られていますが、その要因の一つとして、細菌の細胞膜に存在する多剤排出トランスポーターと呼ばれる膜蛋白質が大きな役割を担っています。多剤排出トランスポーターは、細胞にとって有害な様々な化学物質を能動的に細胞外へ排出する働きをもつ膜蛋白質です。
院内感染などの主要因である多剤耐性緑膿菌などで見られるRND型と呼ばれるタイプの多剤排出トランスポーターでは、村上聡氏(現東京工業大学教授)らにより大腸菌由来のAcrBについて原子レベルの結晶構造が2006年に決定されました。それによると、AcrBは約1000アミノ酸からなる巨大な蛋白質が3個集まった状態(ホモ3量体)で機能しています。そして、各蛋白質は、それぞれ薬剤の取り込み(取込型)、結合(結合型)、排出(排出型)を担う3つの異なる立体構造をとり、(図1A)、細胞内外のプロトン濃度(pH)の差を利用した細胞内へのプロトン移動により得られる駆動力で、各立体構造間を順に変化することで薬剤排出の機能を果たしています(機能的回転メカニズム、図1B)。また、膜貫通部位に存在する3つの荷電アミノ酸(Asp407,Asp408, Lys940)が細胞内へのプロトン移動に寄与しており、排出型の状態のみLys940の側鎖の構造が異なります(図1C)。このことから、プロトン移動によるこれらの荷電アミノ酸のプロトン化状態の変化が引き金となって機能的回転メカニズムを生じていると考えられています。
AcrBは,私たち分子スケールチームの共通の研究ターゲットとして、様々な手法での分子シミュレーションが行われています。私たちの用いた全原子分子動力学法では、生体内の環境をより現実的に考慮するため、細胞膜を構成する脂質分子や水分子およびイオンなどについて水素原子までを含めた系を計算対象としています(図2中央)。そのため、全粒子数が約470,000と非常に大きな系に対する計算となりますが、系内の様々な相互作用を原子レベルで詳細に捉えることができます。そのような全原子分子動力学シミュレーションから、以下のことが明らかになりました。
(1) |
排出型の構造において、Asp407およびAsp408を同時に脱プロトン化した状態では、Lys940の側鎖が結合型および取込型で見られる構造に変化することが分かりました。(図2右パネル)。一方で、Asp408のみをプロトン化させた状態では、排出型の構造を安定に保持できることが分かりました。 |
(2) |
排出型のAsp407、Asp408を同時に脱プロトン化しLys940側鎖の構造が変化したシミュレーションでは、排出型では通常閉じている薬剤の取り込み口が開き、取込型への構造変化の途中の様子が観察されました(図2左パネル)。 |
現在、排出型のLys940の側鎖の構造変化がどのようにして、薬剤の取り込み口が開く構造変化につながるのか、そのメカニズムの解明を目指して研究を進めています。
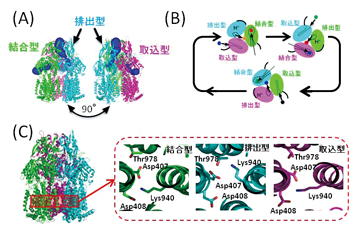 |
図1:多剤排出トランスポーターAcrBの構造と薬剤排出のメカニズム |
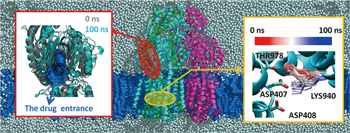 |
図2:全原子分子動力学シミュレーションに用いた系(中央、図中で水分子はグレー、脂質は青の球で示してある。)および結果(左右のパネル)。 |
BioSupercomputing Newsletter Vol.6
- SPECIAL INTERVIEW
- 新しい流体構造連成解析手法(ZZ-EFSI)の開発によっていち早く高い演算性能を達成
東京大学大学院工学系研究科 特任准教授 杉山 和靖 - 開発・高度化チームに聞く「京」の実力と高い性能を引き出すために続くチューニングの取り組み
理化学研究所 生命システム研究センター 生命モデリングコア計算分子設計研究グループ
グループディレクター 泰地 真弘人
理化学研究所 次世代計算科学研究開発プログラム 次世代生命体統合シミュレーション研究推進グループ
生命体基盤ソフトウェア開発・高度化チーム 上級研究員 大野 洋介
理化学研究所 HPCI計算生命科学推進プログラム 高度化推進グループ
高度化推進チーム 上級研究員 小山 洋
理化学研究所 次世代計算科学研究開発プログラム 次世代生命体統合シミュレーション研究推進グループ
生命体基盤ソフトウェア開発・高度化チーム 研究員 舛本 現
理化学研究所 次世代計算科学研究開発プログラム 次世代生命体統合シミュレーション研究推進グループ
生命体基盤ソフトウェア開発・高度化チーム リサーチアソシエイト 長谷川 亜樹
- 研究報告
- 全原子分子動力学シミュレーションによる多剤排出トランスポーターAcrBの機能解析
横浜市立大学大学院生命ナノシステム科学研究科 山根 努 / 池口 満徳( 分子スケールWG) - ヒト循環器系のマルチスケールモデリング
理化学研究所 次世代計算科学研究開発プログラム 梁 夫友( 臓器全身スケールWG) - 初期サッカード視覚運動系のスパイキングニューロンレベルでのモデル化
京都大学 ジャン・モーレン
奈良先端科学技術大学院大学 柴田 智広
沖縄科学技術大学院大学 銅谷 賢治
(脳神経系WG) - 大規模並列用MDコアプログラムの開発
理化学研究所 次世代計算科学研究開発プログラム 大野 洋介(開発・高度化T)
- SPECIAL INTERVIEW
- 複雑な生命現象の理解と予測に向けて計算生命科学の明日を拓く
理化学研究所 HPCI計算生命科学推進プログラム プログラムディレクター 柳田 敏雄
理化学研究所 HPCI計算生命科学推進プログラム 副プログラムディレクター 木寺 詔紀
理化学研究所 HPCI計算生命科学推進プログラム 副プログラムディレクター 江口 至洋
- 研究報告
- 全原子モデルにもとづくヌクレオソームポジション変化の自由エネルギープロファイル計算
日本原子力研究開発機構 量子ビーム応用研究部門
河野 秀俊 / 石田 恒 / 米谷 佳晃 / 池部 仁善
(分野1-課題1) - 骨格筋の活動の推定と脊髄反射の神経モデル
東京大学情報理工学系研究科 中村 仁彦(分野1-課題3)
- 報告
- ISLiM 成果報告会2011
理化学研究所 次世代計算科学研究開発プログラム 田村 栄悦 - 高等学校で行った計算生命科学の授業
理化学研究所 HPCI計算生命科学推進プログラム 鎌田 知佐 / 藤原 康広 / 江口 至洋
