 HPCI戦略プログラム 分野1 予測する生命科学・医療および創薬基盤
HPCI戦略プログラム 分野1 予測する生命科学・医療および創薬基盤
課題1 細胞内分子ダイナミクスのシミュレーション
「京」を用いた大規模シミュレーションによって
細胞内分子ダイナミクスの理解と予測を実現する

理化学研究所 基幹研究所 杉田理論分子科学研究室 主任研究員
杉田 有治
(課題1 代表)
●研究開発課題の全体像
生命科学におけるシミュレーション研究は、その方法論やアルゴリズムの開発の急速な発展に伴い、現在、最もアクティブな研究分野のひとつになっています。ゲノム情報、立体構造情報、細胞内でのタンパク質発現情報などの実験データが次々に生産されているなか、今後、さらにその重要性は高まっていくはずです。また、これまで、生命科学はデータ重視の学問でしたが、これからはそのデータを統合して、生命体システムとしての理解を深める時期に来ています。その意味でも「京」の優れた計算能力によって実現するシミュレーションが、生命科学を予測や制御可能な新しい研究体系に変えていくために果たす役割は大きいといえます。
こうした状況のもと、HPCI戦略プログラム(戦略分野1)「予測する生命科学・医療および創薬基盤」研究がスタートしました。私たちが取り組む課題(細胞内分子ダイナミクスのシミュレーション)では、細胞環境を強く意識した分子・細胞スケールの大規模シミュレーションを行うことによって、細胞内分子ダイナミクスの理解と予測を実現することをめざしています。
これまでも生体分子シミュレーションは数多く行われてきましたが、そのほとんどが、水溶液中あるいは脂質二重膜中におけるタンパク質やDNAなどの振る舞いを明らかにしようとするものでした。しかし、細胞内にあるタンパク質の数と細胞の大きさから計算すると細胞質内の環境が水溶液中の環境と大きく異なっていることは間違いなく、また、それは実験的にも明らかにされつつあります。ひとつのタンパク質が働く現場のすぐ近くには他のタンパク質などが複数存在しているわけです。そのような環境(細胞内分子混雑環境)が、タンパク質の構造やその安定性、そして機能にどのように影響しているのかはまだ完全に理解されているわけではありません。理論的には、タンパク質分子をひとつの粒子に近似したモデルを用いた計算や混み合い効果を統計力学的に調べた研究はあるものの、タンパク質とその周囲の環境を全原子レベルで取り扱った計算事例はほとんど存在していません。私たちは、今回のプロジェクトで、これまでにないスケールで細胞環境に近い条件でタンパク質の働く姿をとらえたいと考えています。
プロジェクトでは、次の3つのサブ課題を実施しています。ひとつは、一分子粒度シミュレーションと分子動力学計算の連携によって新たな研究分野を開拓する「細胞環境下での信号伝達経路のシミュレーション」、長時間の分子動力学計算に基づく自由エネルギー計算によって定量性・予測性を持つ分子シミュレーション達成をめざす「膜タンパク質による細胞膜を隔てた物質輸送」、さらに、核内DNAタンパク質の相互作用を全原子および粗視化分子動力学計算を用いて解析する「核内DNAタンパク質の動的構造と機能の解明」、この3つです。
生命現象を理解し、予測に結び付けていくためには、分子レベルの計算とシステムズバイオロジーを融合させることによって、「細胞まるごと」を理解することが欠かせません。そのため、プロジェクトの終盤では、研究開発によって得られた知見を活用して、「細胞まるごとシミュレーション」をめざした次の課題を明確にしたいと考えています。
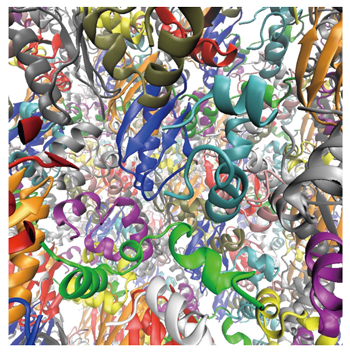
分子レベルの計算から「細胞まるごと」の理解への過程で、「細胞内分子ダイナミクス」の理解と予測は非常に重要な役割を持つ。
 |
 |
|
イオンポンプの生体膜中での分子ダイナミクスをシミュレーションによって見ていくためには、タンパク質、生体膜、水やイオンの約26万におよぶ原子数とともに、その分子間相互作用を含む膨大な計算が必要になる。 |
タンパク質濃度を変化させたときのシミュレーション。細胞質内でタンパク質分子などが混み合った状態を分子シミュレーションで再現し、水分子に与える影響が明らかにされた。 |
●分子シミュレーション研究の目標
課題1「細胞内分子ダイナミクスの理解と予測」を実現するためには、二つの要素が必要です。ひとつは、タンパク質や核酸などの生体分子の“かなり遅い運動”のシミュレーションを実現させること。もうひとつは、分子スケールの研究(原子粒度)と細胞スケールの研究(分子粒度)とを接続し、細胞機能を分子の視点から明らかにしていくことです。
前者については、ミリ秒スケールの大規模な構造変化のシミュレーションに挑みたいと考えています。“遅い”といいましたが、それは私たち研究者の感覚で、実際にはものすごく速い分子運動です。しかし、現在行われている分子シミュレーション研究ではマイクロ秒程度が長時間ダイナミクスの時間スケールなので、その1,000倍も長い、ミリ秒の分子運動は遥かに“遅い”わけです。「京」が完成するまでに私たちが利用可能な計算環境は、約100TFLOPS程度でしたが、「京」の誕生によりおよそ100倍(「京」の演算性能は約10PFLOPS(10,000TFLOPS))強化されました。したがって、これまでと同じような計算機の利用方法で1,000倍長い計算を行うのは、実はかなり難しい数字です。そこで計算手法を何とか工夫して、ミリ秒相当のダイナミクスが見られるようにしようというわけです。
そのための高度な並列化技術の開発をすでに進めています。例えば、多次元レプリカ交換分子動力学計算法というアルゴリズムがあります。この手法ではレプリカと呼ばれる系のコピーについて異なる温度や異なるパラメタでの分子動力学計算を並列に実行し、ある頻度で温度やパラメタを交換することで計算を加速することができます。1個の分子動力学計算について数万CPUを用いた並列化を行うのは難しいのですが、レプリカ交換法では各レプリカの分子動力学計算を数百から数千CPUを用いて並列化し、レプリカを数十から数百用意することで、数万CPUを同時に効率よく利用することが可能になります。こうした方法をいろいろ積み重ねていかなければなりません。
これまでにない計算性能を持つ「京」を活用していくとはいえ、これまで説明したように、計算だけではどうしても限界があります。そのため、この研究課題を実施していくためには、実験とのコラボレーションが欠かせないと考えています。例えば、私たちは立体構造を扱っているので、X線結晶構造解析などによって立体構造が得られれば、それを分子動力学計算するというのがいちばん手っ取り早いわけです。また、NMR(核磁気共鳴)によるスペクトルは溶液中や細胞質中でのタンパク質のダイナミクスに関する情報を与えてくれます。私たちがミリ秒にこだわるのも、それが実現すれば、実験と直接比較ができるのではないかと考えているからです。実験の測定限界とシミュレーションがミリ秒でマッチし、実時間の実験と計算の時間スケールが一致するわけです。
●分子ダイナミクスシミュレーションの成果
「京」の計算性能を効率よく活用していくためのソフトウェア開発や高度化を進める一方で、シミュレーション研究そのものも、すでにいくつも実施されており、成果も得られています。私たちが中心になって進めている細胞質内の分子混雑環境を考慮したシミュレーションもそのひとつです。水を露に含む全原子分子動力学計算を行うことによって、細胞質内と同じ程度に混み合う環境ではタンパク質の周囲に存在する水分子の性質が大きく異なっていることが明らかになっています。それにより、タンパク質間の相互作用も変化し、溶液中と異なるふるまいをしている可能性も示唆されました。細胞内環境での物理パラメタは、実験的に測定することは困難であるため、この計算結果は非常に有用であるといえます。また、今後は「京」を活用することによって、より大規模な系での分子混雑の解析が実現することになり、さらなる研究の進展が期待されます。
また、膜輸送体の分子シミュレーションなども進歩しています。従来はX線結晶構造が解明されてから、分子シミュレーションによるダイナミクスを解明することができるようになるまでしばらく時間がかかっていましたが、最近はX線結晶構造が解けてすぐに脂質二重膜も含めた分子動力学計算が実行できるようになってきました。すでに膜輸送体の輸送サイクルの一部で起こる大規模構造変化がシミュレーションを行うことにより、解明されつつあります。「京」を本格的に用いることにより、膜輸送体が行う膜を隔てた物質輸送をシミュレーションにより明らかにできる日も遠くないと期待しています。
私たちが取り組んでいるプロジェクトには、基礎科学としてだけでなく、創薬や医療への貢献が期待されていることはいうまでもありません。ただ、そうした成果が得られるようになるまでには、まだ時間が必要でしょう。それをめざす一方で、今、私たちが「京」を用いた研究開発で大切にしなければいけないのは、これまでの計算技術では全く不可能であった生命現象のシミュレーションにチャレンジし、その成果を積み上げていくことであろうと思います。それにより、計算科学を用いた生命科学を志す研究者や学生を増やし、彼らが活躍できる場をつくっていく、それも長期的に見れば、このプロジェクトのひとつの役割なのだろうと考えます。
BioSupercomputing Newsletter Vol.7
- SPECIAL INTERVIEW
- 「京」開発担当者に聞く今後のスパコン戦略とエクサスケールに向けた取り組み
富士通株式会社 テクニカルコンピューティングソリューション事業本部
エグゼクティブ・アーキテクト 奥田 基 - 「京」への展開で、実利用に向けた最適化とさらなる規模の拡張を進める大規模仮想ライブラリ
東京大学大学院工学系研究科 化学システム工学専攻 教授 船津 公人
- 研究報告
- 水の誘電率計算から得られる古くて新しい問題
大阪大学蛋白質研究所 中村 春木(分子スケールWG) - 大規模並列計算用流体・構造連成解析プログラムの開発
理化学研究所 情報基盤センター 杉山 和靖(臓器全身スケールWG) - スーパーコンピュータを用いた大規模遺伝子ネットワーク推定ソフトウェア SiGN
東京大学大学院情報理工学系研究科 玉田 嘉紀(データ解析融合WG) - ISLiM研究開発ソフトウェアのソース・コード公開に向けた活動
理化学研究所 次世代計算科学研究開発プログラム 田村 栄悦
- SPECIAL INTERVIEW
- 「京」を用いた大規模シミュレーションによって細胞内分子ダイナミクスの理解と予測を実現する
理化学研究所 基幹研究所 杉田理論分子科学研究室 主任研究員 杉田 有治(課題1 代表) - 日本の優れたコンピュータ技術を活かして革新的な分子動力学創薬に挑戦
東京大学 先端科学技術研究センター 特任教授 藤谷 秀章(課題2 代表)
- 報告
- 大学の新入生に行った計算生命科学の講義
理化学研究所 HPCI計算生命科学推進プログラム 鎌田 知佐
- 体制
- 計算科学技術推進体制
